這一篇是 Mohamed Shoukri 在 2003 年在 SUGI 28 發表的一篇關於用 SAS 檢測兩種不同測量方法差異的文章。在某些跟 bio 相關的研究領域中,通常會有不同的測量方式來針對某一種特殊的計量。每一種測量方式當然都各有其發展的時空背景和優缺點,但使用的人總希望這些測量方式對結果來說不會造成太大的差異。最簡單最直覺的方法就是用 T-test 和 Pearson correlation,但陸續有學者發現這並不適用所有的情況,Altman 和 Bland 分別在 1983 年和 1986 年發表兩篇論文來指出一些問題,並提出新的統計方法來檢定。
Bland 和 Altman 的基本概念是去算兩種不同方法所測出的差異(Vi=Xi-Yi),然後去繪製其和兩種方法的平均值(Ui=(Xi+Yi)/2)的散佈圖。如果描繪點都在零附近打轉的話,那就表示兩種方法應該沒太大的偏差。至於比較有系統的統計檢定方法來測兩種是否有顯著不同,則根據 Shukla 在 1973 年發表的方法,利用 t 分配來檢定下面這個值:
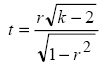
上述的值服從自由度為 k-2 的 t 分配,而 r 是 X 和 Y 的相關係數。
此外,V 的上下限可用下列公式來表示:
Upper Limit(V) = mean(V) + 2*SD(V)
Lower Limit(V) = mean(V) - 2*SD(V)
Shoukri 利用 Ontario Veterinary College 裡的一份資料來跑這個分析。共有下列四個變數:
slide = 鑑定樣本(15 個)
rater = 臨床醫師(2 人)
reading = 醫師判讀鑑定樣本的次數(2 次)
count = 量測結果
由此可知,每位醫生判讀十五份鑑定樣本,每份鑑定樣本都被判讀兩次。
THE TRANSPOSE PROCEDURE
第一步驟需要將資料從單變量架構轉置成多變量架構。也就是從:
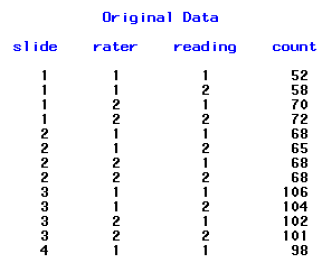
轉置成
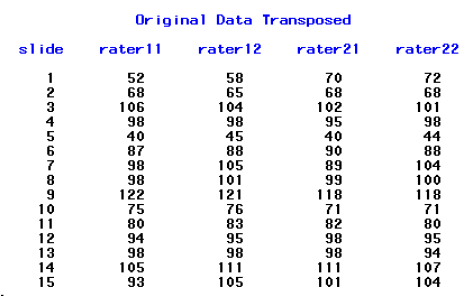
可利用 PROC TRANSPOSE 程序來完成,如下所示:
proc transpose data=rater out=ratertrans (rename=(rater1=rater11 rater2=rater12 rater3=rater21 rater4=rater22) drop=_NAME_) prefix=rater;
var count;
by slide;
run;接著從轉置後的資料來計算不同醫生對兩次判讀結果的平均值以便計算 U 和 V。程式如下:
data newrater;
set ratertrans;
* 1. Compute the averages of the readings made by each rater;
x = (rater11 + rater12)/2;
y = (rater21 + rater22)/2;
* 2. Compute the sum and the difference of the averages of the readings;
u = x + y;
v = x - y;
run;THE CORR PROCEDURE
接著,就可以用 PROC CORR 來計算 U 和 V 的相關係數。結果如下:
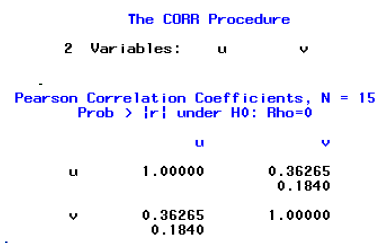
報表顯示相關係數是 0.362,p-value 是 0.184 大於 0.05 顯著水準。因此可以解釋為兩次的判讀有相當程度的準確性。
THE MEANS PROCEDURE
為了要計算 V 的上下限,可以用 PROC MEANS 加上一個 Data procedure 來完成:
proc means data=newrater;
var v;
output mean=meanv std=sdv out=reflines;
run;
data lines;
set reflines;
* 1. Compute the upper and lower limits of v;
up = meanv + (2*sdv);
lo = meanv - (2*sdv);
* 2. Convert the variables, up and lo, into macro variables;
call symput('upper',up);
call symput('lower',lo);
run;特別一提的是,Data procedure 內的 symput 指令是將 upper 和 lower 變數轉成 macro 變數並重新命名為 up 和 lo。
THE GPLOT PROCEDURE
最後,使用 PROC GPLOT 指令把點和上下限描繪出來就大功告成了。
goptions reset=all;
symbol1 v=plus c=blue;
proc gplot data=newrater;
plot v*u / vref=&upper &lower lvref=2 cvref=red;
run;
quit;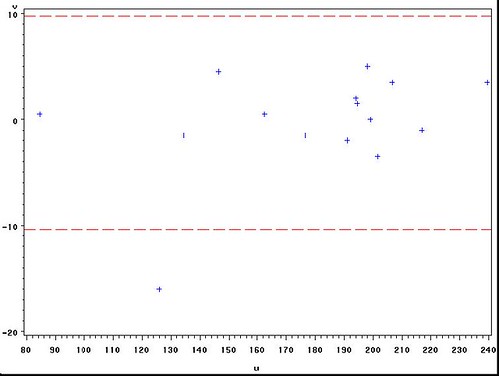
CONCLUSION
一、一般來說,如果有點超出來上下限,就可以判定是離群值。
二、如果(U,V)沒有特殊的趨勢,可以解釋兩個醫生的判讀具有一致性。
三、如果(U.V)有特殊趨勢,則可能要將原始資料做轉換,然後重新跑上述步驟。通常 log-transform 就可以滿足。
CONTACT INFORMATION
Your comments and questions are valued and encouraged.
Contact the author at:
Samia Hashim
King Faisal Specialist Hospital & Research Center
Biostatistics, Epidemiology & Scientific Computing Department
MBC 03-BESC
PO Box 3554
Riyadh 11211
KSA
Work Phone: (+9661) 464-7272 Ext. 39229
Fax: (+9661) 442-4542
Email: samia@kfshrc.edu.sa
Website: www.kfshrc.edu.sa
Mohamed Shoukri
King Faisal Specialist Hospital & Research Center
Biostatistics, Epidemiology & Scientific Computing Department
MBC 03-BESC
PO Box 3554
Riyadh 11211
KSA
Work Phone: (+9661) 464-7272 Ext. 32526
Fax: (+9661) 442-4542
Email: shoukri@kfshrc.edu.sa
Website: www.kfshrc.edu.sa




 連輸出結果都很像:
連輸出結果都很像:








